Chegamos ao terceiro artigo da série sobre NGS. No final preparei uma seleção de artigos científicos para termos sempre disponíveis em nosso dia a dia.
Complementações e comentários serão bem-vindos!
Por que macrodissecar a área tumoral antes do NGS?
Como definir a porcentagem de células tumorais para o estudo genômico?
Conforme falamos nos artigos anteriores, o estudo das mutações somáticas é feito preferencialmente no tecido tumoral que foi fixado em formol e emblocado em parafina. A sensibilidade do sequenciamento é drasticamente maior quando fazemos a extração apenas do DNA tumoral.
Mas, por que não posso simplesmente cortar o bloco de parafina e sequenciar?
Veja esse exemplo:
Essa foto é parte do meu acervo de casos que foram sequenciados durante meu mestrado. Observe como o tumor representa uma pequena área da lâmina, e que a maioria do tecido é um parênquima sem alterações. Se cortamos esse bloco e levarmos diretamente para a extração, teremos o DNA tumoral e o DNA das outras células não neoplásicas misturados.
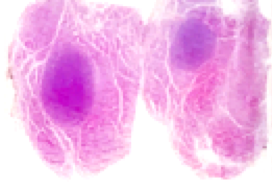
Estaríamos gerando uma “contaminação” com DNA não neoplásico e isso diminuirá a sensibilidade da detecção das mutações. Uma forma simples de entender, seria pensar que ao extrair todo o conteúdo da lâmina, sem macrodissecar, estaríamos “diluindo” o DNA tumoral antes de leva-lo ao sequenciamento.
Entendida a importância de extrairmos apenas o DNA tumoral isolado, os passos realizados pelo patologista nessa etapa são:
– delimitar na lâmina a área tumoral, determinar a porcentagem de células neoplásicas (PCN) dentro da área delimitada, verificar a representatividade da amostra (tamanho), e por fim confirmar o diagnóstico feito previamente.
Uma das principais causas de falha nos testes moleculares é devido a baixa porcentagem de células tumorais. Essa porcentagem também é crucial para a interpretação do resultado molecular; ela é tão importante que é considerado uma boa prática informa-la no laudo. Testar amostras com porcentagem tumoral insuficiente pode gerar resultados falso negativos, já que não é possível afirmar se o tecido realmente não tem a mutação ou se ela era insuficiente para ser detectada.
Os principais estudos sugerem um valor mínimo de 20% de celularidade tumoral para um boa confiança do sequenciamento, mas essa porcentagem pode variar de acordo com o teste.
A porcentagem de células tumorais também tem um impacto na interpretação das métricas de qualidade do NGS, pois amostras com baixa celularidade precisam de uma cobertura maior para serem aprovadas. Além disso a correta estimativa da % ajuda a distinguir as variantes somáticas das germinativas, com implicações para o manejo do paciente e de seus parentes.
Lembrete importante: mutação somática é aquela que está presente SOMENTE nas células tumorais, por isso sua frequência alélica é baixa, estando em torno de 5 a 15 %. Mutação germinativa é aquela que está presente em TODAS as células do paciente, por isso sua frequência alélica será alta, variando em torno de 50% até 100%.
Agora, mãos à obra!
Vou descrever como tudo isso acontece na prática.
- O primeiro passo é selecionar a lâmina mais representativa do tumor e com uma caneta permanente, selecionar a área com a maior densidade de núcleos neoplásicos viáveis e com a menor densidade de células inflamatórias. É importante salientar aqui que é a porcentagem de núcleos que está sendo estimada e não a área ocupada pela neoplasia, ou ainda o tamanho dos núcleos.
- Evite áreas de necrose, áreas com muitas células inflamatórias, mucina e adipócitos. Evite selecionar estroma desmoplásico. É melhor ter uma pequena área, mas que seja homogênea, do que selecionar uma grande área com muitas células não neoplásicas de permeio.
- Em seguida, demarcamos as lâminas não coradas, que serão raspadas na sala de extração. Esses cortes não corados devem ser grossos, com 10 micrômetros cada; podemos utilizar de 5 até mais de 10 lâminas dessas não coradas; tudo depende do tamanho do tumor.
- A raspagem a seco das lâminas não coradas é a maneira mais utilizada pelos laboratórios para macrodissecar um tecido tumoral. Os fragmentos raspados da área delimitada são colocados em um microtubo e seguem para a extração de DNA.
Para ilustrar, veja abaixo esse caso de neoplasia maligna ovariana, que foi enviada para teste molecular. O tecido apresentava focos de necrose, por isso selecionei áreas descontínuas, mas que apresentavam maior uniformidade. As duas lâminas da direita são os cortes não corados, que também foram marcados com caneta para guiar a raspagem do material.

Na prática, quando precisamos sequenciar uma amostra muito pequena ou com baixa porcentagem tumoral, é essencial adicionar ao laudo final uma nota informando ao oncologista que, nesses casos, um resultado negativo não exclui a presença de variantes, visto que a amostra apresentava baixa celularidade. Mediante o contexto clínico do paciente, o médico solicitante pode enviar outros blocos da neoplasia para tentarmos obter uma lâmina mais representativa.
Espero que o texto tenha ajudado!
Ainda assim, selecionei os principais artigos científicos para quem precisar se aprofundar nesse assunto:
1.Neoplastic cell percentage estimation in tissue samples for molecular oncology: recommendations from a modified Delphi study. Dufraing K, van Krieken JH, De Hertogh G, Hoefler G, Oniscu A, Kuhlmann TP, Weichert W, Marchiò C, Ristimäki A, Ryška A, Scoazec JY, Dequeker E. Histopathology. 2019 Sep;75(3):312-319. doi: 10.1111/his.13891. Epub 2019 Jul 18. PMID: 31054167; PMCID: PMC6851675.
2.Implementing NGS-based BRCA tumour tissue testing in FFPE ovarian carcinoma specimens: hints from a real-life experience within the framework of expert recommendations. Rivera D, Paudice M, Gismondi V, Anselmi G, Vellone VG, Varesco L; Ligurian BRCA Working Group. J Clin Pathol. 2021 Sep;74(9):596-603. doi: 10.1136/jclinpath-2020-206840. Epub 2020 Sep 7. PMID: 32895300
3.Comparison of Next-Generation Sequencing, Quantitative PCR, and Sanger Sequencing for Mutation Profiling of EGFR, KRAS, PIK3CA and BRAF in Clinical Lung Tumors. Gao J, Wu H, Shi X, Huo Z, Zhang J, Liang Z. Clin Lab. 2016;62(4):689-96. doi:10.7754/clin.lab.2015.150837. PMID: 27215089
4.Next-Generation Sequencing Analysis of Laser-Microdissected Formalin-Fixed and Paraffin-Embedded (FFPE) Tissue Specimens. Mägel L, Bartels S, Lehmann U. Methods Mol Biol. 2018;1723:111-118. doi: 10.1007/978-1-4939-7558-7_5. PMID: 29344856
5.Simultaneous detection of BRCA mutations and large genomic rearrangements in germline DNA and FFPE tumor samples. Enyedi MZ, Jaksa G, Pintér L, Sükösd F, Gyuris Z, Hajdu A, Határvölgyi E, Priskin K, Haracska L. Oncotarget. 2016 Sep 20;7(38):61845-61859. doi: 10.18632/oncotarget.11259. PMID: 27533253 Free PMC article.
6.Testing of mutations in BRCA1 and BRCA2 genes in tumor tissues – possibilities and limitations]. Vošmiková H, Ryška A, Sieglová K, Laco J. Cesk Patol. 2016 Fall;52(4):210-214. PMID: 27869447 Review. Czech.
7.Pre-Analytical Considerations for Successful Next-Generation Sequencing (NGS): Challenges and Opportunities for Formalin-Fixed and Paraffin-Embedded Tumor Tissue (FFPE) Samples. Arreaza G, Qiu P, Pang L, Albright A, Hong LZ, Marton MJ, Levitan D. Int J Mol Sci. 2016 Sep 20;17(9):1579. doi: 10.3390/ijms17091579. PMID: 27657050 Free PMC article.
8.Comparison of targeted next-generation sequencing (NGS) and real-time PCR in the detection of EGFR, KRAS, and BRAF mutations on formalin-fixed, paraffin-embedded tumor material of non-small cell lung carcinoma-superiority of NGS. Tuononen K, Mäki-Nevala S, Sarhadi VK, Wirtanen A, Rönty M, Salmenkivi K, Andrews JM, Telaranta-Keerie AI, Hannula S, Lagström S, Ellonen P, Knuuttila A, Knuutila S. Genes Chromosomes Cancer. 2013 May;52(5):503-11. doi: 10.1002/gcc.22047. Epub 2013 Jan 30. PMID: 23362162.
Até breve!
Dra. Ana Carolina Paniza
Patologista Molecular
CRM-SP 151630